library(tidyverse)4 Transformación de tablas y otros
4.1 Objetivos de la sesión
- En esta clase exploraremos algunas funciones para manipulación de tablas tanto de dplyr como del paquete tidyr.
4.2 Base de datos
Abrimos nuestra base de datos:
library(readxl)
dataf<-read_xlsx("data/resultados_sv_2021.xlsx")Exploramos:
names(dataf)[1] "cod_dep_reniec" "cod_prov_reniec" "REGION" "PROVINCIA"
[5] "cod_prov_inei" "castillo" "keiko" 4.3 Configuración de variables
Vemos que la variable castillo y keiko se encuentran como character.
glimpse(dataf)Rows: 196
Columns: 7
$ cod_dep_reniec <chr> "01", "01", "01", "01", "01", "01", "01", "02", "02", …
$ cod_prov_reniec <chr> "0101", "0102", "0103", "0104", "0105", "0106", "0107"…
$ REGION <chr> "AMAZONAS", "AMAZONAS", "AMAZONAS", "AMAZONAS", "AMAZO…
$ PROVINCIA <chr> "CHACHAPOYAS", "BAGUA", "BONGARA", "LUYA", "RODRIGUEZ …
$ cod_prov_inei <chr> "0101", "0102", "0103", "0105", "0106", "0104", "0107"…
$ castillo <chr> "55.15", "65.544", "57.451", "56.686", "53.112", "92.1…
$ keiko <chr> "36.763", "28.8", "35.737", "37.088", "38.672", "4.59"…Este caso podemos coercionar estos vectores para que sean numéricos utilizando la función as.numeric():
as.numeric(dataf$castillo) #En este caso SÓLO ESTAMOS VISUALIZANDO la variable convertida a numérica [1] 55.150 65.544 57.451 56.686 53.112 92.161 61.776 58.977 58.935 61.926
[11] 61.942 38.930 56.688 43.794 75.240 76.341 67.635 70.180 69.701 44.603
[21] 69.261 61.586 81.195 70.801 70.047 43.059 47.049 69.789 75.732 78.336
[31] 79.447 84.269 81.510 79.215 59.327 84.377 54.287 41.808 71.520 76.045
[41] 67.556 79.215 78.386 84.310 76.028 73.638 69.137 77.637 84.040 87.936
[51] 77.697 70.904 73.889 54.418 50.932 75.934 57.308 68.935 81.736 84.258
[61] 66.050 68.532 68.640 74.852 67.928 74.899 66.851 88.161 83.051 82.460
[71] 91.364 87.578 91.693 87.341 0.000 86.659 88.315 85.377 82.743 84.016
[81] 82.842 83.765 64.469 80.839 64.721 0.000 59.452 62.896 75.751 77.521
[91] 75.799 50.644 76.736 57.070 80.801 82.187 86.792 44.609 43.780 45.341
[101] 46.986 50.010 56.084 51.852 47.903 63.186 47.891 63.272 52.641 49.755
[111] 64.179 31.193 56.630 45.825 47.916 39.507 52.822 51.573 36.770 42.964
[121] 51.974 29.757 51.421 37.399 45.856 40.274 32.261 42.030 49.562 42.818
[131] 43.395 50.248 57.267 40.582 41.221 52.420 39.994 59.098 54.044 39.226
[141] 52.899 25.861 0.000 0.000 65.978 0.000 58.509 74.556 80.779 58.032
[151] 71.090 81.404 34.417 33.274 66.050 68.005 39.255 38.913 26.121 39.899
[161] 37.140 80.898 92.249 84.934 90.296 90.997 87.792 89.261 85.170 79.809
[171] 87.709 89.659 91.065 90.279 52.660 53.294 49.506 51.882 56.660 43.038
[181] 60.099 62.580 49.943 55.339 67.644 82.742 79.789 88.103 34.070 26.044
[191] 25.527 30.353 40.919 65.022 41.578 0.000dataf$castillo<-as.numeric(dataf$castillo) #Con el signo de asignación estamos CREANDO (o sobreescribiendo) la variable.Lo comprobamos solicitando su clase:
class(dataf$castillo)[1] "numeric"Si queremos hacer muchas coerciones de forma simultánea podemos utilizar la función lapply(), la cual es una de las funciones de la familia “apply” en R, diseñada para aplicar una función a los elementos de una lista o a los componentes de un objeto.
dataf[,6:7]<-lapply(dataf[,6:7], as.numeric)4.4 Etiquetas a las variables
El etiquetado de columnas en R sirve para asignar descripciones más significativas o detalladas a las variables de un conjunto de datos. Las etiquetas mejoran la legibilidad y comprensión del código y los resultados, y son particularmente valiosas en análisis estadísticos, informes, y visualizaciones donde el contexto y la claridad son cruciales.
Por ejemplo, label(dataf$castillo) <- “Votación por Castillo” asigna la etiqueta “Votación por Castillo” a la columna castillo del dataframe dataf. Este método es útil para aclarar el significado de las variables, especialmente cuando los nombres de las columnas son breves o técnicos.
library(Hmisc)
label(dataf$castillo)<-"Votación por Castillo"
label(dataf$keiko)<-"Votación por Keiko"4.5 Repasa case_when()
Recuerda que la función case_when() del paquete dplyr sirve para recodificar datos y crear nuevas variables o modificar variables existentes basándose en múltiples condiciones.
Permite evaluar varias condiciones utilizando una sintaxis similar a una instrucción “if-else”. Esta función es particularmente útil cuando necesitamos recodificar una variable en varias categorías o cuando tenemos múltiples condiciones a evaluar.
Podemos realizar:
dataf<-dataf |> # Data
select(3,4,6,7) |> # Selecciono estas columnas
mutate(nivel_respaldo_castillo= #Creo una nueva variable que se llamará "nivel_respado_castillo"...
case_when(castillo<30~"Bajo", # donde se le asigna la etiqueta "Bajo" si es menor a 30...
castillo<60~"Medio", # la etiqueta "Medio" si es menor a 60 y...
TRUE~"Alto")) # la etiqueta "Alto" a todas las demás que no cumplen con lo anterior. Como en este caso la variable es ordinal, hay que especificarle ello al R:
dataf$nivel_respaldo_castillo<-factor(dataf$nivel_respaldo_castillo,
levels = c("Bajo", "Medio", "Alto"), ordered = T)
label(dataf$nivel_respaldo_castillo) = "Nivel de respaldo a Pedro Castillo"Podríamos generar otra variable que indique quién ganó en esa provincia. Cómo sería?
4.6 Tablas de resumen con gtsummary

El paquete gtsummary en R (2019) es una herramienta diseñada para simplificar y agilizar la creación de tablas resumen estadísticas y de datos. Es conocido por ser útil especialmente en el contexto de la investigación biomédica y la epidemiología, pero aplica para todos los rubros.
Este paquete permite a los usuarios generar rápidamente tablas bien formateadas que resumen características básicas de un conjunto de datos, como medias, medianas, intervalos, y frecuencias. También es capaz de realizar pruebas estadísticas y presentar sus resultados de manera clara y concisa. La gran ventaja de gtsummary es que convierte procesos que normalmente requerirían varias líneas de código y un conocimiento profundo de estadísticas en tareas mucho más sencillas y directas, facilitando la comunicación de resultados estadísticos y la elaboración de informes y publicaciones científicas.
4.6.1 tbl_summary() para resumir
Se utiliza para crear tablas resumen detalladas y bien formateadas de las características de las variables en un conjunto de datos.
Para esta función se le pueden incluir los siguientes “tipos” de variables: continuous, categorical, dichotomous, y continuous2.
Las variables continuous se refieren a variables numéricas que se resumen en una sola fila, típicamente con estadísticas como la media y la desviación estándar.
Las variables categorical son aquellas con un número limitado de categorías o grupos, y se resumen mostrando conteos y porcentajes para cada categoría.
Las variables dichotomous son un caso especial de variables categóricas con solo dos categorías, como ‘Sí’ o ‘No’.
Finalmente, continuous2 es similar a continuous, pero ofrece un resumen más detallado al desglosar las estadísticas en dos o más filas, permitiendo una representación más completa de los datos numéricos.
Antes de hacer nuestras tablas, hay que decirle al R que el lenguaje que estamos usando es español y que nuestro separador de decimales es el “.”:
library(gtsummary)#StandWithUkrainetheme_gtsummary_language(
language = "es",
decimal.mark = "."
)Setting theme `language: es`Primero hagamos una prueba con nuestra data que tiene dos variables continuous y una categorical. Podemos notar que para la primera sólo lo resume en una línea (mediana y rango intercuartílico), mientras que para la segunda te muestra la frecuencia y porcentaje de cada categoría.
dataf |>
select(castillo, keiko, nivel_respaldo_castillo) |>
tbl_summary()| Característica | N = 1961 |
|---|---|
| Votación por Castillo | 63 (48, 79) |
| Votación por Keiko | 29 (14, 43) |
| Nivel de respaldo a Pedro Castillo | |
| Bajo | 11 (5.6%) |
| Medio | 81 (41%) |
| Alto | 104 (53%) |
| 1 Mediana (RIQ); n (%) | |
Podemos modificar para que ahora nos muestre la Media(Desviación estándar) para todas las numéricas:
library(gtsummary)
dataf |>
select(castillo, keiko, nivel_respaldo_castillo) |>
tbl_summary(statistic = list(all_continuous() ~ "{mean} ({sd})"))| Característica | N = 1961 |
|---|---|
| Votación por Castillo | 61 (21) |
| Votación por Keiko | 30 (18) |
| Nivel de respaldo a Pedro Castillo | |
| Bajo | 11 (5.6%) |
| Medio | 81 (41%) |
| Alto | 104 (53%) |
| 1 Media (DE); n (%) | |
Ahora generamos una variable dicotómica dummy que indique si es que en la provincia ganó Castillo:
dataf<-dataf |> # Data
mutate(castillo_gano=
case_when(castillo>keiko~1,
TRUE~0))
label(dataf$castillo_gano)="Ganó Pedro Castillo"Y solicitamos nuevamente la tabla resumen. Vemos que al ser dicotómica lo presenta en una sola línea:
dataf |>
select(castillo, keiko, nivel_respaldo_castillo, castillo_gano) |>
tbl_summary(statistic = list(all_continuous() ~ "{mean} ({sd})"))| Característica | N = 1961 |
|---|---|
| Votación por Castillo | 61 (21) |
| Votación por Keiko | 30 (18) |
| Nivel de respaldo a Pedro Castillo | |
| Bajo | 11 (5.6%) |
| Medio | 81 (41%) |
| Alto | 104 (53%) |
| Ganó Pedro Castillo | 149 (76%) |
| 1 Media (DE); n (%) | |
Y qué pasaría si queremos convertir una de nuestra continuous en continuous2? Es decir, solicitarle que nos de mayor información?
dataf |>
select(castillo, keiko, nivel_respaldo_castillo, castillo_gano) |>
tbl_summary(
type = keiko ~ "continuous2",
statistic = list(all_continuous() ~ "{mean} ({sd})",
all_continuous2() ~ c("{mean} ({sd})", "{min}, {max}")))| Característica | N = 1961 |
|---|---|
| Votación por Castillo | 61 (21) |
| Votación por Keiko | |
| Media (DE) | 30 (18) |
| Rango | 0, 69 |
| Nivel de respaldo a Pedro Castillo | |
| Bajo | 11 (5.6%) |
| Medio | 81 (41%) |
| Alto | 104 (53%) |
| Ganó Pedro Castillo | 149 (76%) |
| 1 Media (DE); n (%) | |
4.6.2 Personalización de encabezados
Finalmente, podemos personalizar aún más la tabla con los comandos modify_header y modify_caption para editar los encabezados en nuestro idioma.
dataf |>
select(castillo, keiko, nivel_respaldo_castillo, castillo_gano) |>
tbl_summary(
type = keiko ~ "continuous2",
statistic = list(all_continuous() ~ "{mean} ({sd})",
all_continuous2() ~ c("{mean} ({sd})", "{min}, {max}"))) |>
modify_header(label = "**Variable de interés**") |>
modify_caption("**Tabla resumen con `gtsummary`**") | Variable de interés | N = 1961 |
|---|---|
| Votación por Castillo | 61 (21) |
| Votación por Keiko | |
| Media (DE) | 30 (18) |
| Rango | 0, 69 |
| Nivel de respaldo a Pedro Castillo | |
| Bajo | 11 (5.6%) |
| Medio | 81 (41%) |
| Alto | 104 (53%) |
| Ganó Pedro Castillo | 149 (76%) |
| 1 Media (DE); n (%) | |
4.6.3 Videos recomendados
Te recomiendo ver estos dos tutoriales sobre gtsummary, ambos muy buenos. Ten en cuenta que en estos casos ya entran más a detalle sobre la generación de tablas de resumen con este paquete:
https://www.youtube.com/watch?v=6QTrzd2Wxrs
https://www.youtube.com/watch?v=tANo9E1SYJE
4.7 Mutating Joins
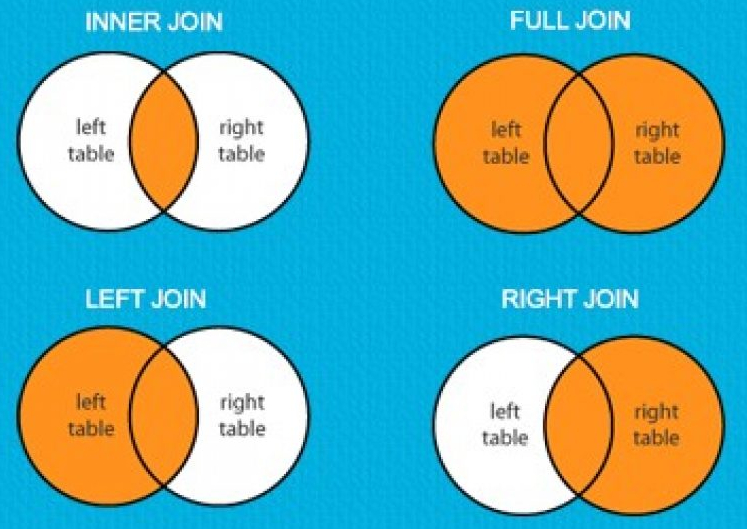
Los mutating joins son una serie de funciones en R, particularmente en el paquete dplyr, que permiten combinar dos tablas basadas en columnas clave compartidas. Estas funciones no solo agregan filas de una tabla a otra, sino que también modifican las columnas de la tabla original al añadir información de la tabla complementaria. Entre los mutating joins más comunes se encuentran left_join(), right_join(), inner_join() y full_join(), cada uno diseñado para combinar tablas de diferentes maneras según las relaciones entre las claves de las tablas.
4.7.1 Tablas de ejemplo
Generamos dos tablas a modo de ejemplo:
# Crear la primera tabla de ejemplo
tabla1 <- data.frame(
ID = 1:10,
Nombre = c('Ana', 'Carlos', 'Elena', 'Luis', 'María', 'Pedro', 'Rosa', 'José', 'Clara', 'Marta'),
Edad = c(23, 34, 45, 28, 39, 41, 30, 38, 33, 29)
)
# Crear la segunda tabla de ejemplo
tabla2 <- data.frame(
ID = c(1, 3, 5, 7, 9, 11, 12, 13, 14, 15),
Puntaje = c(88, 92, 75, 85, 90, 67, 72, 83, 94, 77)
)Las visualizamos:
tabla1 ID Nombre Edad
1 1 Ana 23
2 2 Carlos 34
3 3 Elena 45
4 4 Luis 28
5 5 María 39
6 6 Pedro 41
7 7 Rosa 30
8 8 José 38
9 9 Clara 33
10 10 Marta 29La tabla 2:
tabla2 ID Puntaje
1 1 88
2 3 92
3 5 75
4 7 85
5 9 90
6 11 67
7 12 72
8 13 83
9 14 94
10 15 774.7.2 Variable KEY
La variable key es la columna o conjunto de columnas que existen en ambas tablas y que se usan para encontrar las filas correspondientes en cada tabla. Es esencialmente el criterio de emparejamiento. Por ejemplo, si tienes una tabla con datos demográficos de personas (tabla1) y otra con sus puntajes en un examen (tabla2), la clave podría ser una columna como ID que identifique de manera única a cada persona en ambas tablas.
La variable key (o clave) se precisará el argumento by dentro de las funciones left_join(), right_join(), inner_join() y full_join().
4.7.3 left_join()
left_join() toma todas las filas de tabla1 (la tabla de la izquierda) y añade las columnas de tabla2 donde haya coincidencias en la columna ID. Si no hay coincidencia en tabla2, las nuevas columnas tendrán valores NA
left_join_result <- left_join(tabla1, tabla2, by = "ID")
left_join_result ID Nombre Edad Puntaje
1 1 Ana 23 88
2 2 Carlos 34 NA
3 3 Elena 45 92
4 4 Luis 28 NA
5 5 María 39 75
6 6 Pedro 41 NA
7 7 Rosa 30 85
8 8 José 38 NA
9 9 Clara 33 90
10 10 Marta 29 NA4.7.4 right_join()
right_join() toma todas las filas de tabla2 (la tabla de la derecha) y añade las columnas de tabla1 donde haya coincidencias en la columna ID. Si no hay coincidencia en tabla1, las nuevas columnas tendrán valores NA.
right_join_result <- right_join(tabla1, tabla2, by = "ID")
right_join_result ID Nombre Edad Puntaje
1 1 Ana 23 88
2 3 Elena 45 92
3 5 María 39 75
4 7 Rosa 30 85
5 9 Clara 33 90
6 11 <NA> NA 67
7 12 <NA> NA 72
8 13 <NA> NA 83
9 14 <NA> NA 94
10 15 <NA> NA 774.7.5 inner_join()
Devuelve solo las filas donde haya coincidencias en la columna ID en ambas tablas. Las filas sin coincidencia son eliminadas del resultado.
inner_join_result <- inner_join(tabla1, tabla2, by = "ID")
inner_join_result ID Nombre Edad Puntaje
1 1 Ana 23 88
2 3 Elena 45 92
3 5 María 39 75
4 7 Rosa 30 85
5 9 Clara 33 904.7.6 full_join()
Devuelve todas las filas de ambas tablas. Si hay coincidencia en la columna ID, las filas se combinan; si no hay coincidencia, se incluyen las filas con valores NA en las columnas donde no hubo coincidencia.
full_join_result <- full_join(tabla1, tabla2, by = "ID")
full_join_result ID Nombre Edad Puntaje
1 1 Ana 23 88
2 2 Carlos 34 NA
3 3 Elena 45 92
4 4 Luis 28 NA
5 5 María 39 75
6 6 Pedro 41 NA
7 7 Rosa 30 85
8 8 José 38 NA
9 9 Clara 33 90
10 10 Marta 29 NA
11 11 <NA> NA 67
12 12 <NA> NA 72
13 13 <NA> NA 83
14 14 <NA> NA 94
15 15 <NA> NA 774.7.7 Lectura recomendada
Te sugiero leer el capítulo “Joins” de R for Data Science en este link.